Mise en évidence d’un problème d’annotation automatique
1. Le gène HAP1 dans les bases de données de référence
Lorsque nous recherchons le gène HAP1 chez C. glabrata dans la CGD (base de données de référence pour les levures Candida), nous obtenons :
Standard Name : HAP1
Systematic Name : CAGL0K05841g
Alias : ZCF27
Description : Has domain(s) with predicted DNA binding, DNA-binding transcription factor activity, RNA polymerase II-specific, zinc ion binding activity and role in regulation of transcription, DNA-templated, transcription, DNA-templated
Ortholog(s) in non-CGD species : S. cerevisiae (HAP1)
Lorsque nous recherchons CAGL0K05841g dans GRYC (seconde base de référence pour C. glabrata), aucune donnée n’est disponible.
Si nous utilisons l’identifiant utilisé dans nos puces à ADN basé sur GRYC (CAGL0B03421g), nous obtenons dans la CGD :
Standard Name : pas d’information
Systematic Name : CAGL0B03421g
Alias : ZCF4
Description : Ortholog(s) have DNA-binding transcription activator activity, RNA polymerase II-specific, RNA polymerase II cis-regulatory region sequence-specific DNA binding activity
Ortholog(s) in non-CGD species : S. cerevisiae (HAP1)
| Dans la base de données GRYC, nous avons comme description « similar to uniprot | P12351 Saccharomyces cerevisiae YLR256w HAP1 transcription factor ». |
En résumé, pour un même gène HAP1, nous avons deux gènes possibles dans la CGD dont un seul est décrit dans GRYC.
2. Et au niveau des séquences nucléotidiques ?
Nous avons ensuite récupéré la séquence nucléotidique de HAP1 sur la SGD. À l’aide de BLAST (Basic Local Alignment Search Tool)1, nous avons recherché les séquences qui présentaient le même alignement. Les deux gènes les plus significatifs sont ceux décrits ci-dessous. Les résultats du BLAST sont regroupés dans le Tableau 1.
| 1 |
CAGL0B03421g |
| 2 |
CAGL0K05841g |
| ... | ... |
Enfin, nous avons réalisé un alignement de la séquence du gène CAGL0B03421g présent dans GRYC. Nous obtenons avec BLAST un seul résultat chez C. glabrata présenté dans le Tableau 2.
CAGL0K05841g
Candida glabrata CBS138 HAP1 Has domain(s) with predicted DNA binding, DNA-binding transcription factor activity, RNA polymerase II-specific,
zinc ion binding activity and role in regulation of transcription, DNA-templated, transcription, DNA-templated
Length = 4,119
Score = 136 (123 bits), Expect = 2e-27
Identities = 185/263 (70.3%), Positives = 185/263 (70.3%), Frame = +1 / +1
Nous avons donc deux gènes extrêmement proches ayant bénéficié d’un transfert d’annotation de S. cerevisiae. Comment pourrions-nous décider entre les deux ?
3. Une solution basée sur la synténie
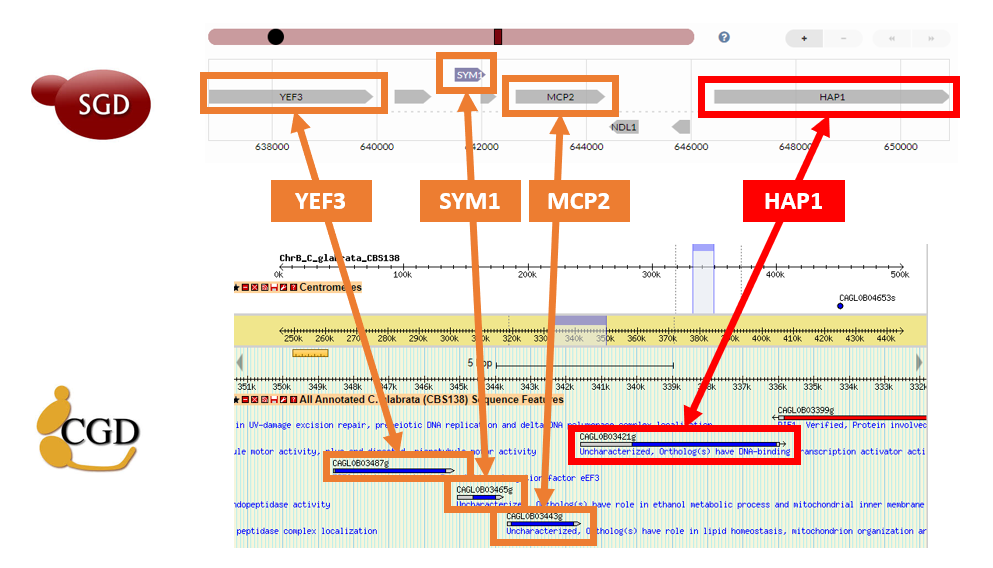
Pour sélectionner un des deux gènes, nous avons décidé d’utiliser la synténie. Nous avons donc recherché si les gènes qui entourent notre gène d’intérêt sont les mêmes par orthologie chez S. cerevisiae. Les 3 gènes qui précèdent CAGL0B03421g ont leurs orthologues qui précèdent HAP1 chez S. cerevisiae (Figure 1). Il n’existe pas de synténie pour le gène CAGL0K05841g. En résumé, l’annotation proposée pour le gène HAP1 dans la CGD semble erronée et la cause la plus probable est une annotation automatique par transfert non vérifiée.
-
Outil permettant de trouver des régions de similarité locale entre les séquences ↩