La Gene Ontology
1. Une ontologie
En informatique, une ontologie est un ensemble de termes définis qui sont structurés entre eux. Il s’agit d’un modèle de données qui définit des relations entre des termes dans un domaine donné.
2. La Gene Ontology
La Gene Ontology est un projet de bio-informatique qui a pour objectif de définir une structure entre les descriptions des gènes et dérivés. Pour faciliter son organisation, cette ontologie est basée sur un vocabulaire contrôlé c’est-à-dire un ensemble de termes prédéfinis et sélectionnés pour optimiser la recherche d’information. Cette base de données est organisée comme un graphe orienté acyclique (DAG). Il s’agit d’un graphe orienté qui ne possède pas de circuit et qui peut être considéré comme une hiérarchie. Chaque terme est en relation avec un ou plusieurs termes. Sa particularité est que les termes qui la compose sont applicables à tous les organismes (multicellulaires, unicellulaires, eucaryotes, procaryotes, …).
3. Les GO terms
Il s’agit des termes que nous retrouvons dans cette ontologie. Ils s’organisent sous 3 grands domaines :
- Les composants cellulaires (Cellular Component) : surface cellulaire, membrane plasmique, etc. ;
- Les fonctions moléculaires (Molecular function) : liaison ionique, activité de transport, etc. ;
- Les processus biologiques (Biological Process) : pathogenèse, croissance du symbiote chez l’hôte, etc.
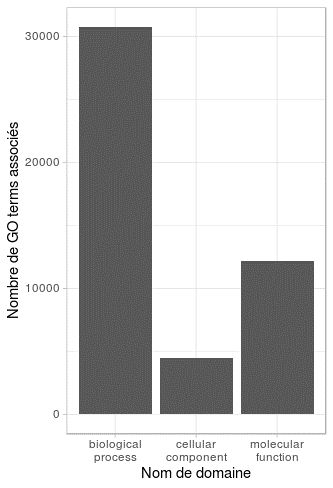
Le domaine bilogical process regroupe plus de 30000 GO terms, suivi par molecular function avec plus de 12000 GO terms et enfin cellular component avec plus de 4000 Go terms (Figure 120).
Chaque GO term est défini au minimum par :
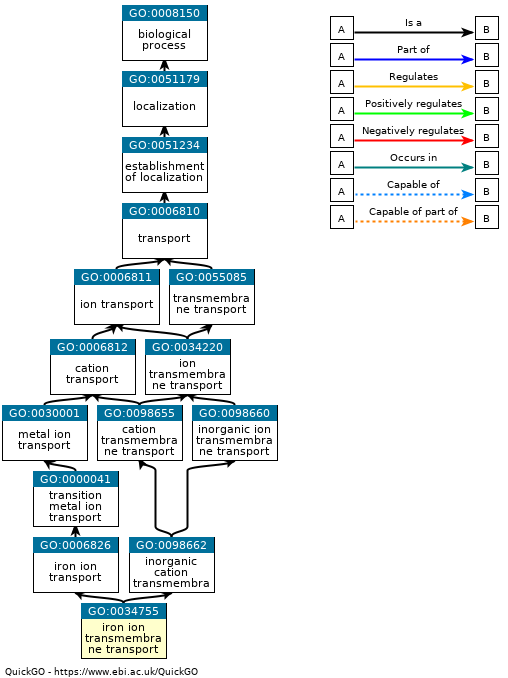
- Un identifiant unique alphanumérique : GO:0034755 ;
- Un nom : iron ion transmembrane transport ;
- Une définition avec ses sources citées : A process in which an iron ion is transported from one side of a membrane to the other by means of some agent such as a transporter or pore. PMID:11390404
- Le domaine auquel il appartient : Biological Process
D’autres informations peuvent aussi être associées comme des synonymes, des références, …. Toutes ces informations sont organisées et diffusées sur le site QuickGO. Il est possible de retrouver notamment le DAG contenant le GO term recherché comme celui proposé à la Figure 121.
L’ensemble du vocabulaire est stocké dans des fichiers au format .obo (Open Biomedical Ontologies). Un extrait est proposé ci-dessous (Extrait 1)
format-version: 1.2
data-version: releases/2020-01-01
subsetdef: gocheck_do_not_annotate "Term not to be used for direct annotation"
subsetdef: gocheck_do_not_manually_annotate "Term not to be used for direct manual annotation"
subsetdef: goslim_agr "AGR slim"
subsetdef: goslim_aspergillus "Aspergillus GO slim"
subsetdef: goslim_candida "Candida GO slim"
subsetdef: goslim_chembl "ChEMBL protein targets summary"
subsetdef: goslim_drosophila "Drosophila GO slim"
subsetdef: goslim_flybase_ribbon "FlyBase Drosophila GO ribbon slim"
subsetdef: goslim_generic "Generic GO slim"
subsetdef: goslim_metagenomics "Metagenomics GO slim"
subsetdef: goslim_mouse "Mouse GO slim"
subsetdef: goslim_pir "PIR GO slim"
subsetdef: goslim_plant "Plant GO slim"
subsetdef: goslim_pombe "Fission yeast GO slim"
subsetdef: goslim_synapse "synapse GO slim"
subsetdef: goslim_yeast "Yeast GO slim"
synonymtypedef: syngo_official_label "label approved by the SynGO project"
synonymtypedef: systematic_synonym "Systematic synonym" EXACT
default-namespace: gene_ontology
remark: cvs version: use data-version
remark: Includes Ontology(OntologyID(OntologyIRI(<http://purl.obolibrary.org/obo/go/never_in_taxon.owl>))) [Axioms: 18 Logical Axioms: 0]
ontology: go
[Term]
id: GO:0000001
name: mitochondrion inheritance
namespace: biological_process
def: "The distribution of mitochondria, including the mitochondrial genome, into daughter cells after mitosis or meiosis, mediated by interactions between mitochondria and the cytoskeleton." [GOC:mcc, PMID:10873824, PMID:11389764]
synonym: "mitochondrial inheritance" EXACT []
is_a: GO:0048308 ! organelle inheritance
is_a: GO:0048311 ! mitochondrion distribution
[Term]
id: GO:0000002
name: mitochondrial genome maintenance
namespace: biological_process
def: "The maintenance of the structure and integrity of the mitochondrial genome; includes replication and segregation of the mitochondrial chromosome." [GOC:ai, GOC:vw]
is_a: GO:0007005 ! mitochondrion organization